Professional Documents
Culture Documents
Manual VMD
Uploaded by
Evelyn Ruales Dávila100%(1)100% found this document useful (1 vote)
707 views11 pagesCopyright
© © All Rights Reserved
Available Formats
PDF, TXT or read online from Scribd
Share this document
Did you find this document useful?
Is this content inappropriate?
Report this DocumentCopyright:
© All Rights Reserved
Available Formats
Download as PDF, TXT or read online from Scribd
100%(1)100% found this document useful (1 vote)
707 views11 pagesManual VMD
Uploaded by
Evelyn Ruales DávilaCopyright:
© All Rights Reserved
Available Formats
Download as PDF, TXT or read online from Scribd
You are on page 1of 11
Instituto de Qumica * Facultad de Ciencias
Avenida Universidad 330 * Valparaso * Chile
Fono (56-32) 2274994
waldo.acevedo@ucv.cl * http://institutoquimica.ucv.cl
OBJETIVOS
Preparacin del sistema con visualizador VMD (Visual Molecular Dynamics)
Solvatacin del sistema
Aprender acerca de archivo input y output de NAMD para correr simulacin molecular
Aprender los pasos bsicos de la simulacin Molecular mediante el uso de la herramienta NAMD
Introduccin
DINMICA MOLECULAR
Este laboratorio consiste en el aprendizaje de pasos bsicos de Dinmica Molecular, las cuales son:
PRCTICA 11: DINMICA MOLECULAR
Preparacin del sistema
Minimizacin
Equilibracin
Antes de correr nuestra simulacin molecular, NAMD requiere 5 archivos importantes:
Un archivo PDB (Protein Data Bank), contiene las coordenadas atmicas y / o las velocidades para
el sistema. Los archivos PDB puede ser generados manualmente, u obtenido por va Internet
http://www.pdb.org
Un archivo PSF (Protein Structure File), contiene la informacin estructural de la protena
Un archivo de topologa (*.top o Force File Topology File), contiene la informacin sobre tipo de
tomos, cargas y cmo los tomos estn conectado en una molcula. Nota que el archivo PDB
contiene slo las coordenadas, pero no la informacin de conectividad
Un archivo de parmetro (*.par o Force Field Parameter File). Un campo de fuerza es una
expresin matemtica que indica el potencial del sistema. CHARMM, AMBER, y GROMACS son tres
tipos de campo de fuerza y NAMD es apto para usar todos ellos. El archivo de parmetro define
longitud de enlace, longitudes del equilibrio, etc
Un archivo de configuracin, en la cual el usuario especifica todas las opciones que NAMD debiera
usar para correr la simulacin. El archivo de configuracin le dice a NAMD cmo la simulacin es
corrida
Construya el rbol de archivo
En este caso, vamos a enfocarnos en el estudio de
la ubiquitina (PDB: 1UBQ), una pequea protena
de 76 aminocidos encontrada clulas eucaritica,
cuya funcin es marcar otra protena, tal como
receptores de membrana. Para su destruccin
procesos conocido como proteasoma
home
usuario
escritorio
1-1-build
1-2-esphere
1-3-box
SECCIN 1: PREPARACIN DEL SISTEMA
GENERACIN DEL ARCHIVO PSF (PROTEIN STRUCTURE FILE)
PASO 1: Vamos al directorio /1-1-build y encontrars los archivos requeridos para construir nuestro archivo
PSF:
archivo PDB de la ubiquitina, PDB: 1UBQ, es tpicamente obtenido a travs del Protein Data Bank
archivos de parmetros (par_all27_prot_lipid.inp )
topologa (top_all27_prot_lipid.inp).
En la consola tipear VMD y cargar la protena ubiquitina (1UBQ.pdb)
File New Molecule...
Nota: La estructura rayos-X obtenida de Protein Data Bank no contiene los tomos de hidrgenos de la
ubiquitina. Esto es porque la cristalografa de rayos-X usualmente no resuelve tomos de hidrgenos, El
archivo PDB que generars con psfgen, junto con el PSF contendrn coordenadas para tomos de hidrgenos
de la estructura. De esta forma, en la minimizacin de la energa de la protena garantizar que sus posiciones
sean razonables.
PASO 2: Elegir Extensions Tk Console . Asegrate de que ests en el directorio /1-1-build
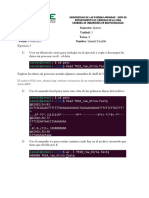
En la consola Tk, tipea los siguientes comandos
set ubq [atomselect top protein]
$ubq writepdb ubqp.pdb
Nota: En el paso anterior slo haz creado el archivo ubqp.pdb, contiene las coordenadas de la ubiquitina sin los
hidrgenos
PASO 3: Ahora crearemos el archivo PSF de la ubiquitina.
El paquete psfgen de VMD es muy til para construir el archivo psf. En la lnea de comando tipea gedit
build_protein.tcl. Tipea la siguiente lnea de comando:
package require psfgen
topology top all27 prot lipid.inp
pdbalias residue HIS HSE
pdbalias atom ILE CD1 CD
segment U {pdb ubqp.pdb}
coordpdb ubqp.pdb U
guesscoord
writepdb ubq.pdb
writepsf ubq.psf
Despus de tipear, guarda el archivo File Save. Asegrate que ests en el directorio /1-1-build. Luego, sales
del editor de texto cliqueando FileExit
El archivo que has creado contiene los comandos necesarios para crear el archivo PSF de la ubiquitina con los
tomos de hidrgenos y sin agua
Lnea 1: Ests corriendo psfgen con VMD. Esta lnea requiere que el paquete psfgen est
disponible para ser llamado por VMD
Lnea 2: Carga el archivo de topologa top_all27_prot_lipid.inp
Lnea 3: Cambia el nombre de residuo histidina por el nombre apropiado encontrado en el archivo
de topologa. HSE es uno de los 3 nombres basado en el estado protonacin de la cadena lateral.
Lnea 4: El tomo nombrado CD1 (carbono ) en el residuo isoleucina es llamado CD, su
nombre adecuado en el archivo de topologa.
Lnea 5: Un segmento llamado U es creado, contiene todos los tomos de ubqp.pdb El comando
segment aade todos los tomos de hidrgenos
Lnea 6: Coordenadas son ledos desde ubqp.pdb, residuos y nombre se tomos son acertados. Las
etiquetas del antiguo segmento ser anuladas
Lnea 7: Coordenadas de tomos faltantes (hidrgenos) son aadidos basados en la definicin de
residuos del archivo de topologa
Lnea 8: Nuevo archivo pdb con las coordenadas completas de todos los tomos, incluyendo los
hidrgenos.
Lnea 9: Archivo psf con la informacin completa de la estructura de la protena
En la consola tcl/tk escribe la siguiente lnea de comando.
play buil_protein.tcl
PASO 4 Una vez construido el psf, revisaremos que los archivos pdb y psf de la ubiquitina vamos a cargar el
sistema.
Copie el arhivo ubq.pdb y ubq.psf al directorio /1-2-sphere y 1-3-box.
SECCIN 2: SOLVATACIN DE LA PROTENA
Ahora, la protena necesita ser solvatada. Puedes hacerle de dos formas, localizando la protena en:
Una esfera de agua, en preparacin de minimizacin y equilibrado sin condiciones peridicas de
borde.
Una caja de agua, en preparacin de minimizacin y equilibrado con condiciones peridicas de
borde.
1.b.1 Ubiquitina en una esfera de agua
PASO 1. Situarse en el directorio /1-2-sphere. encontrars un script tcl wat_sphere.tcl para crear la esfera de
agua. En la consola tipear VMD y cargar la protena ubiquitina (ubq.pdb y ubq.psf)
vmd ubq.psf ubq.pdb
PASO2. Luego llamars al script, sto ubicar la ubiquitina en una esfera de agua lo ms pequea posible. Elegir
Extensions Tk Console . En la consola Tk, tipea los siguientes comandos
play wat_sphere.tcl
PASO 3. Los outputs generado por el script wat_sphere.tcl estar el centro y el radio de la esfera de agua.
Registrar estos nmeros, los necesitars ms tarde. El script tendrs creado los archivos ubq_ws.pdb y
ubq_ws.psf, respectivamente, ubiquitina en una esfera de agua. Estos estarn en el directorio /1-2-sphere.
Revisa esto tipeando ls
Luego cargar las coordenadas que estn en el archivo pdb, Cliquear File New Molecule , en la ventana
principal de VMD. En el Molecule File Browser, usa el botn Browse para encontrar el archivo ubq_ws.pdb .
Cargar presionando el botn Load
Una vez revisada vamos sal ir , abrimos la tk consola y tipeamos exit.
1.b.2 Ubiquitina en una caja de agua
Construir Automticamente
Vamos a cargar los archivos generados.
PASO 1. Cargar las coordenadas que estn en el archivo pdb. Cliquear File New Molecule , en la ventana
principal de VMD. En el Molecule File Browser, usa el botn Browse para encontrar el archivo ubq.pdb .
Cargar presionando el botn Load
O bien puede tipear por consola:
vmd ubq.pdb
PASO 2. En la ventana Tk Consola, tipear:
set ubq [atomselect top all]
measure minmax $ubq
Esto analiza todos los tomos del sistema y entrega los valores mnimos y mximos de coordenadas x, y, z del
sistema protena-agua. Estos valores tambin definen origen del sistema de coordenada. El centro de la caja
de agua puede ser determinado determinando el punto medio de cada uno de los tres lados. Por ejemplo, si los
valores mnimos y mximo del eje X la caja sera 10.44 y 51.12, respectivamente. Entonces el punto medio:
51.12 + 10.44 = 30.78
2
PASO 3. Cliquear Extensiones Modeling Add Solvation Box, se mostrar lo siguiente:
Una vez definido los parmetros cliquear sobre el botn Solvate, obtienes los archivos con los parmetros de la
protena en una caja de agua
Nombre de arhivo psf
Nombre de arhivo pdb
Nombre de arhivo de salida
Dimensiones de la caja, lo puedes
obtener tipeando en la consola Tk lo
siguiente:
set prot [atomselect top all]
measure minmax $prot
Construir Manualmente
PASO 1. Situarse en el directorio /1-3--box. encontrars un script tcl wat_box.tcl para crear el cubo de agua.
En la consola tipear VMD y cargar la protena ubiquitina (ubq.pdb y ubq.psf)
vmd ubq.psf ubq.pdb
PASO2. Luego llamars al script, sto ubicar la ubiquitina en ula caja de agua lo ms pequea posible. Elegir
Extensions Tk Console . En la consola Tk, tipea los siguientes comandos
play wat_box.tcl
PASO 3. En el script usted ver el siguiente cdigo
package require solvate
solvate ubq.psf ubq.pdb -t 15 -o ubq_wb
El comando package require solvate carga el paquete solvate, asi VMD podr llamarlo. El paquete solvate
colocar la protena en una caja de agua. La opcin t crea las dimensiones de la caja de agua de tal manera
que hay una capa de agua de 15 en cada direccin desde el tomo ms lejano del centro. La opcin o crea
archivos output ubq_ws.pdb y ubq_ws.psf para ubiquitina en caja de agua.
Otra forma de crear una caja de agua es usando la herramienta de vmd.
SECCIN 3: SIMULACIN MOLECULAR
3.1 Ubiquitina en una esfera de Agua
En esta seccin, examinars la minimizacin y equilibrado de la ubiquitina en una esfera de agua localizada en
el vaco
PASO 1. Dirigirse al directorio /1-2-sphere, aqu encontrars el archivo de configuracin llamado
ubq_ws_eq.conf , para la minimizacin y el equilibrado de la ubiquitina en una esfera de agua.
Los archivos de salidas para la minimizacin y el equilibrado de la ubiquitina en una esfera de agua estar
ubicado en este directorio, el archivo de configuracin, es slo un archivo de entrada, que contiene todas las
instrucciones necesarias para correr la simulacin. Adems, encontrars en el directorio /1-2-sphere archivo
pdb (ubq_ws.pdb), psf (ubq_ws.psf) y archivo de parmetros (par_all27_prot_lipid.inp). Podras comprobar
usando el comando ls
Paso Alternativo
1.1 Fijar backbone de la protena usando el script fix_backbone.ucl. Esto se realiza previo a la Minimizacin
del sistema
1.2 Fijar Restraint a los Carbono alfa de los aminocidos usando el script restraint_ca.tcl Esto se realiza previo a
la Equilibrado del sistema
PASO 2. Abrir el archivo de configuracin, ubq_ws_eq.conf , con un editor de texto gedit
El archivo de configuracin puede ser muy simple al principio, pero lo examinaremos lnea por lnea para
determinar su funcin en tu simulacin
La seccin JOB DESCRIPTION
Contiene slo comentario y su propsito es informar al simulador qu tipo de simulacin se reaizar. T
comentario debiera decir:
# Minimization and Equilibration of Ubiquitin in a Water Sphere
La seccin ADJUSTABLE PARAMETERS
Contiene 5 comandos:
1. structure: llama al archivo psf para describir el sistema (ubq_ws.psf)
2. coordinates: llama al archivo pdb que contiene las coordenadas iniciales (ubq_ws.pdb)
3. set temperatura: se crea una variable llamada temperature el cual almacena un valor de
temperatura inicial del sistema. Si localiza el texto $temperature en el archivo de configuracin,
NAMD leer como etiqueta el nmero 310 (corresponde a la temperatura inicial del sistema)
4. set outputname: crea un variable llamada outputname el cual almacena el nombre genrico de los
archivos de salida. Si localizas el texto $outputname en el archivo de configuracin NAMD leer
como etiqueta ubq_ws_eq
5. firsttimestep: asigna un valor numrico para el primer paso de la simulacin. Esto es til cuando
reinicia la simulacin.
La seccin SIMULATION PARAMETERS
Input
paraTypeCharmm: Indica si o no el archivo de parmetro es el formato usado por el
campo de fuerza CHARMM, off indica que esto no lo es
parameters: llama a los parmetros campo de fuerza desde el archivo
par_all27_prot_lipid.inp
temperature: Asigna la temperatura inicial del sistema en Kelvin, con ello se asigna la
velocidad aleatoria a tomos seleccionado de la distribucin de Maxwell tal que se obtiene
promedio de energa cintica dado la temperatura
Force-Field Parameters
exclude: Especifica cules interacciones atmica debe ser excluido. La etiqueta scaled1-4
indica que las interacciones entre los tomos 1 y2 , 1y3 son despreciados y las interacciones
entre los tomos 1 y 4 son debilitados
1-4scaling: especifica el grado de libertad en la cual interaccin entre los tomos 1 y 4 es
tomado en cuenta. Esta variable puede tomar valor hexadecimal 0 (falso) o 1 (verdadero)
cutoff: indica la distancia en ngstrom el cual las interacciones electrosttica y Van der Waals
son cortados
switching: indica si la funcin switching es usada sin problema tomando las interacciones
electrosttica y Van der Waals desde 0 a una distancia cutoff
switchdist: indica la distancia en ngstrom el cual la forma funcional de interacciones
electrosttica y Van der Waals son modificadas para permitir que sus valores se acerquen a
cero a una distancia cutoff
pairlistdist: diseado para hacer que el clculo se ms rpido
Integrator Parameters
timestep: indica el valor time step usado en la simulacin
rigidBonds: especifica cules enlaces formados por hidrgenos con considerados como
rgidos (no vibracin). El valor all indica que todos los enlaces formados por hidrgenos y
algn otro tomos.
nonbondedFreq: especifica el nmero de time steps en que las interacciones no enlzantes
son calculadas.
fullElectFrequency: especifica el nmero de time steps en que las interacciones
electrosttica son calculadas.
stepspercycle: Atomos que son reasignados en lista de pares una vez por ciclo.
Constant Temperature Control
langevin: indica s o no la simulacin usa la dinmica Langevine, usa valores off u on.
langevinDamping: asigna valores de acoplamiento de Langevin. Cuantifica la afliccin
aplicada al sistema, removiendo energa desde el sistema, etc.
langevinTemp: Dinmica de Langevine puede ser aplicada a todos los tomos o a slo
tomos no-hidrgeno del sistema. Este comando especifica la temperatura a la cual se
mantiene estos tomos, incluyendo friccin y la fuerza que actuar en ellos
langevinHydrogen: indica s o no la dinmica Langevine ser aplicado a tomos de
hidrgenos en la simulacin, usa valores off u on.
Output
outputName: Muchos tipos de datos de salida son escrito por NAMD en la simulacin. Este
comando especifica el prefijo para archivos de salida. NAMD retorna dos archivos salida por
cada simulacin: archivo pdb que contiene las coordenadas finales de todos los tomos del
sistema y un archivo pdb que contiene las velocidades finales de todos los tomos del
sistema. Las extensiones de estos archivos son: *.coor y *.vel
restartfreq: Durante la simulacin. NAMD puede crear archivo restart, uno el cual es un
archivo pdb que almacena las coordenadas atmica y otro que almacena las velocidades
atmica. Este comando especifica por cada cunto time steps se escriba la salida en el archivo
restart
dcdfreq: El archivo dcd contiene slo coordenadas atmica y stos son escritos a un archivo
muchas veces en el curso de la simulacin
outputEnergies: especifica por cada cunto time steps la energa del sistema es ecrito al
archivo .log
outputPressure: especifica por cada cunto time steps la presin del sistema es ecrito al
archivo .log
La seccin EXTRA PARAMETERS contiene los comandos la cual se aplican a simulaciones ms
especficas. Incluye comando la cual caracteriza condiciones esfricas de borde en una esfera de agua.
Spherical boundary conditions. Los siguientes tres comandos debe ser especificado para
que condiciones esfrica de borde trabaje apropiadamente
sphericalBC: indica si o no la simulacin usa condiciones esfricas de borde, usa valores off u
on.
sphericalBCcenter: asigna las coordenadas x, y, z del centro de la esfera para el cual
condiciones esfricas de borde es aplicada
sphericalBCr1: asigna distancia en ngstrom en el cual el primer potencial de borde acta.
sphericalBCk1: el primer potencial aplicado condiciones esfricas de borde que mantiene las
esferas juntas (o separadas) es armnico. Este comando asigna valores de constante de
fuerza a este potencial
sphericalBCexp1: asigna el valor al exponente usada en la frmula de potencial
La seccin EXECUTION SCRIPT, contiene tres comandos, los primeros dos es aplicado a la
minimizacin y el ltimo aplicado al equilibrado.
Minimizacin
minimize: asigna nmero de iteraciones en el cul los tomos varan sus posiciones
para buscar el mnimo local en el potencial
reinitvels: Minimizacin es ejecutado en el sistema despus de que todas las
velocidades atmicas se les han asignado cero. Este comando reasigna velocidades
atmicas tal que el sistema comienza a la temperatura especificada.
Run: Asigna el nmero de pasos en cul corre el equilibrado de Dinmica Molecular (ene ste
caso 2500 pasos es equivalente a 5000 fs o 5 ps)
PASO 3. Una vez asignados los parmetros, corremos nuestra simulacin con la siguiente lnea de comando.
charmrun ++local +pN namd2 ubq_ws_eq.conf > ubq ws_eq.log &
N: nmero de core
PASO 4. Anlisis
Para hacer el anlisis cargue los siguientes archivos en vmd .
vmd ubq_ws.psf ubq_ws_eq.dcd
Usted puede ver la simulacin molecular:
Luego, para graficar tiempo de simulacin versus energa
Extensions Analysis NAMD Plot
3.2 Ubiquitina en una Caja de Agua: Simulacin en condiciones peridica de borde
En esta seccin, examinars la minimizacin y equilibrado de la ubiquitina en una caja de agua en condiciones
peridica de borde
PASO 1. Dirigirse al directorio /1-3-box, aqu encontrars el archivo de configuracin llamado ubq_wb_eq.conf ,
para la minimizacin y el equilibrado de la ubiquitina en una esfera de agua.
Los archivos de salidas para la minimizacin y el equilibrado de la ubiquitina en una esfera de agua estar
ubicado en este directorio, el archivo de configuracin, es slo un archivo de entrada, que contiene todas las
instrucciones necesarias para correr la simulacin. Adems, encontrars en el directorio /1-3-box archivo pdb
(ubq_wb.pdb), psf (ubq_wb.psf) y archivo de parmetros (par_all27_prot_lipid.inp). Podras comprobar usando
el comando ls
Paso Alternativo
1.1 Fijar backbone de la protena usando el script fix_backbone.ucl. Esto se realiza previo a la Minimizacin
del sistema
1.2 Fijar Restraint a los Carbono alfa de los aminocidos usando el script restraint_ca.tcl Esto se realiza previo a
la Equilibrado del sistema
PASO 2. Abrir el archivo de configuracin, ubq_wb_eq.conf , con un editor de texto gedit
El archivo de configuracin contiene algunos comandos que son diferentes archivo de configuracin de la
esfera de agua
La seccin SIMULATION PARAMETERS, hay tres nuevas categora de parmetros que han sido aadidas.
Periodic Boundary Conditions
Los tres vectores bases de la celda peridica entregan la forma y el tamao de la celda
peridica. Ellos eson cellBasisVector1, cellBasisVector2, and cellBasisVector3.
cellOrigin: especifica las coordenadas del centro de la celda peridica en ngstrom
wrapWater: Este comando es usado junto a condiciones peridicas de borde.
wrapAll: Es lo mismo a wrapWater: pero es usado a todas las molculas
PME (full-system periodic electrostatics)
PME (Particle Mesh Ewald o grilla de partculas) Este mtodo es til para considerar las
interacciones electrostticas en el sistema cuando est presente las condiciones peridicas de borde.
La suma de Ewald es eficiente para el clculo de fuerza en el sistema peridico. La grilla de partculas
es una grilla en 3D creada en el sistema, cuya carga del sistema est distribuida. Desde estas cargas,
potenciales y fuerzas en el sistema es determinada.
PME: indica s o no la simulacin usa el mtodo de suma de Ewald, usa valores yes o no
PMEGridSpacing: Asigna un radio mnimo entre el nmero de puntos de la grilla a lo largo
de cellBasisVector.
Uno puede definir el tamao de la grilla PME manualmente PMEGridSizeX,
PMEGridSizeY, y PMEGridSizeZ. Esto asigna el tamao de la grilla PME cellBasisVector1,
2 y 3, respectivamente
Constant Pressure Control (volumen variable)
useGroupPressure: NAMD calcula la presin del sistema basados en la fuerza entre tomos
y su energa cintica. El comando especfica si la interaccin que implica hidrgenos debera
ser contado para todos los tomos de hidrgenos o simplemente para un grupos de tomos
de hidrgenos, usa valores yes o no. Debes asignar yes si rigidBonds son asignados
useFlexibleCell: Especifica s o no quieres permitir las tres dimensiones de la celda peridica
vara. Usa valores yes o no
useConstantArea: NAMD permite mantener el rea seccional x y sea constante mientras
vara la dimensin z. Usa valores yes o no
langevinPiston: Especifica s o no, la simulacin usa Piston Langevin para controlar la presin
del sistema. Usa valores yes o no
langevinPistonTarget: Indica la presin (en bar) con el cual el prisrton Langevin se mantiene
langevinPistonPeriod: asigna un tiempo constante de oscilacin en fs para piston Langevin
langevinPistonDecay: asigna un tiempo constante de Camping en fs para piston Langevin
langevinPistonTemp: asigna temperatura en K para piston Langevin.
Output
xstFreq: archivo que contiene un registro de los parmetros de la celda peridica. Este
comando especifica por cada cunto time steps la configuracin debe ser registrada.
PASO 3. Una vez asignados los parmetros, corremos nuestra simulacin con la siguiente lnea de comando.
charmrun ++local +pN namd2 ubq_wb_eq.conf > ubq_wb_eq.log &
N: nmero de core
PASO 4. Anlisis
Para hacer el anlisis cargue los siguientes archivos en vmd .
vmd ubq_wb.psf ubq_wb_eq.dcd
Usted puede ver la simulacin molecular:
Luego, para graficar tiempo de simulacin versus energa
Extensions Analysis NAMD Plot
You might also like
- Administración de sistemas operativosFrom EverandAdministración de sistemas operativosRating: 3.5 out of 5 stars3.5/5 (8)
- Mem Tutorial - En.esDocument43 pagesMem Tutorial - En.esViviana AndreaNo ratings yet
- UF0852 - Instalación y actualización de sistemas operativosFrom EverandUF0852 - Instalación y actualización de sistemas operativosRating: 5 out of 5 stars5/5 (1)
- Practica Uso de NAMDDocument8 pagesPractica Uso de NAMDEdEr Aguirre0% (1)
- Iniciación al diseño de circuitos impresos con Altium DesignerFrom EverandIniciación al diseño de circuitos impresos con Altium DesignerNo ratings yet
- Actividad 3.1Document25 pagesActividad 3.1Aj AljnbNo ratings yet
- Taller Eukaryotic Room 3 Ed-Feb-27-2023Document7 pagesTaller Eukaryotic Room 3 Ed-Feb-27-2023dsescobarmNo ratings yet
- Proyecto Openstack SDDocument6 pagesProyecto Openstack SDHernan AvilaNo ratings yet
- Procedimiento Configuracion Software en El Mismo PCDocument2 pagesProcedimiento Configuracion Software en El Mismo PCJhonny MuñozNo ratings yet
- VMD Introduccion PDFDocument12 pagesVMD Introduccion PDFMilton Paredes AvalosNo ratings yet
- ENSAMBLAJE DEL GENOMA DE E. Coli V2 PDFDocument12 pagesENSAMBLAJE DEL GENOMA DE E. Coli V2 PDFMaria MolinaNo ratings yet
- Examen Resuelto UnixDocument81 pagesExamen Resuelto UnixkenyrgNo ratings yet
- InformeDocument11 pagesInformeDavid SilveraNo ratings yet
- Lab. 4.2 - Práctica de Laboratorio Extraer Un Ejecutable de Un PCAPDocument9 pagesLab. 4.2 - Práctica de Laboratorio Extraer Un Ejecutable de Un PCAPNapoleonNo ratings yet
- Capítulo 7Document20 pagesCapítulo 7Marisol Huenuanca FloresNo ratings yet
- GLAB S015 Resolución de ProblemasDocument13 pagesGLAB S015 Resolución de ProblemasAdrian Alexsander Hurtado CruzNo ratings yet
- 12.2.2.10 SergioRaulAparicioDocument8 pages12.2.2.10 SergioRaulAparicioRaúl MuñozNo ratings yet
- 12.2.2.10 Lab - Extract An Executable From A PCAPDocument8 pages12.2.2.10 Lab - Extract An Executable From A PCAPKAREN JULIANA GONZALEZ MARTINEZNo ratings yet
- Unix EssentialsDocument10 pagesUnix EssentialsThomas VélezNo ratings yet
- Manual de Integracion Con JAVADocument24 pagesManual de Integracion Con JAVAEdgar RojasNo ratings yet
- Taller LinuxDocument3 pagesTaller LinuxOscar Mauricio Parra CorreaNo ratings yet
- ProblemasUnix BisDocument81 pagesProblemasUnix Bisprofessor_inf_sucroNo ratings yet
- Taller DockingDocument25 pagesTaller DockingDaniel Bustos GuajardoNo ratings yet
- NqfPbQyETm2vK-IBCcAfiw s4 Taller2 APIDocument5 pagesNqfPbQyETm2vK-IBCcAfiw s4 Taller2 APIjuan sebastian perdomo becerraNo ratings yet
- Práctica - 3.3. - UNIXDocument9 pagesPráctica - 3.3. - UNIXprofesoraemNo ratings yet
- 2C2014 - SO - RáfagaDocument32 pages2C2014 - SO - RáfagaGabriel MureriNo ratings yet
- Laboratorio 11Document20 pagesLaboratorio 11wvaliente2011No ratings yet
- Práctico #0 - Marclé EmilianoDocument18 pagesPráctico #0 - Marclé EmilianoEmiliano MarcléNo ratings yet
- Lab14 RSYNC 2020 IDocument5 pagesLab14 RSYNC 2020 IJean Pierre Charlie Velasquez Espinoza0% (1)
- 113 Ch2 AssignmentDocument10 pages113 Ch2 AssignmentHugo WongNo ratings yet
- UPC Segundo ParcialSistemasDocument3 pagesUPC Segundo ParcialSistemasjeiison realesNo ratings yet
- Ejercicios Resueltos en PascalDocument156 pagesEjercicios Resueltos en Pascalsepa83% (12)
- Laboratorio 5Document2 pagesLaboratorio 5PepeNo ratings yet
- Práctica 0 Arquitectura de Redes IDocument15 pagesPráctica 0 Arquitectura de Redes INatalia Pinto EstebanNo ratings yet
- Tutorial SPICE CaptureDocument65 pagesTutorial SPICE Captureluisfourier10No ratings yet
- Manual de GromacsDocument17 pagesManual de GromacsJuan Jeferson AlosillaNo ratings yet
- Guía DockingDocument13 pagesGuía DockingErickNo ratings yet
- Curso Omnet PDFDocument0 pagesCurso Omnet PDFLiz AyalaNo ratings yet
- 4.recoleccion Datos Volatiles WindowsDocument27 pages4.recoleccion Datos Volatiles WindowsalbeirovegaNo ratings yet
- Ficha Complementaria N 2Document6 pagesFicha Complementaria N 2Gery MorganNo ratings yet
- Lab14 RSYNC 2020 IDocument4 pagesLab14 RSYNC 2020 IJean Pierre Charlie Velasquez EspinozaNo ratings yet
- Temario Cursos OmicasDocument12 pagesTemario Cursos OmicasNini OrtizNo ratings yet
- Actividad Entregable 1Document4 pagesActividad Entregable 1Luis Cebrian AldunateNo ratings yet
- 27.2.10 Lab - Extract An Executable From A PCAP - ILMDocument6 pages27.2.10 Lab - Extract An Executable From A PCAP - ILMJhanssel HolguinNo ratings yet
- Tarea 1U2. Castillo - SamuelDocument11 pagesTarea 1U2. Castillo - Samuelameliacoro09No ratings yet
- 7.6.3 Lab - Automated Testing Using pyATS and GenieDocument21 pages7.6.3 Lab - Automated Testing Using pyATS and GeniehernandezpaloNo ratings yet
- 2.práctica #2.2 2022Document2 pages2.práctica #2.2 2022MariaE RandoNo ratings yet
- Banco de Preguntas-Unidad2 RespuestasDocument7 pagesBanco de Preguntas-Unidad2 RespuestasAlexia GutiérrezNo ratings yet
- Laboratorio 1Document4 pagesLaboratorio 1edu_eddyNo ratings yet
- Sistemas de Control de VersionesDocument14 pagesSistemas de Control de VersionesLuis Miguel Nuñez CastroNo ratings yet
- Docking Agus - GrafenoDocument10 pagesDocking Agus - GrafenoPedro MartinezNo ratings yet
- Taller - Fundamentos de Bioinformática IDocument4 pagesTaller - Fundamentos de Bioinformática IUna Persona CualquieraNo ratings yet
- Práctica Obligatoria: Sistemas Operativos MonopuestoDocument3 pagesPráctica Obligatoria: Sistemas Operativos MonopuestoPacoNo ratings yet
- Aso 2021 02 N2Document4 pagesAso 2021 02 N2Antonio MembriveNo ratings yet
- Examen Práctico PES - MayoDocument6 pagesExamen Práctico PES - Mayojjsc18No ratings yet
- GLAB-S10-MHUACO-2023-01 FabDocument22 pagesGLAB-S10-MHUACO-2023-01 FabPaola Barrera SimborthNo ratings yet
- Ventura SOTR HuberDocument9 pagesVentura SOTR HuberdnetkaizenNo ratings yet
- Comunicacion y Sincronizacion Entre Procesos en LinuxDocument21 pagesComunicacion y Sincronizacion Entre Procesos en LinuxAlejandro Gomez75% (4)
- Practicas de Laboratorio Curso AsteriskDocument75 pagesPracticas de Laboratorio Curso AsteriskEttore PelliccioniNo ratings yet
- Taller 2Document5 pagesTaller 2omniarangoNo ratings yet
- ¿En Qué Consiste La Nivelación Topográfica - Axisima®, Ingeniería y Medio AmbienteDocument8 pages¿En Qué Consiste La Nivelación Topográfica - Axisima®, Ingeniería y Medio AmbienteIng HectorNo ratings yet
- Capitulo VII La Policía AdministrativaDocument2 pagesCapitulo VII La Policía AdministrativaElizabethSaraiNo ratings yet
- Endocrown 2018 PDFDocument3 pagesEndocrown 2018 PDFEstefanía Paz Mardones YáñezNo ratings yet
- Oclusion Funcional-InformeDocument23 pagesOclusion Funcional-InformeJosé Hernández100% (3)
- Implantación de Actividades LudicasDocument3 pagesImplantación de Actividades Ludicasana200886No ratings yet
- Tarea Semana III. Infotecnologia - WordDocument8 pagesTarea Semana III. Infotecnologia - WordYeury FlorentinoNo ratings yet
- Coaching Deportivo PDFDocument14 pagesCoaching Deportivo PDFRafael ChamorroNo ratings yet
- Manejo y Tratamiento de Residuos Químicos en El Laboratorio de Química General de La Universidad Simón Bolívar. Leonarda Carrillo.Document7 pagesManejo y Tratamiento de Residuos Químicos en El Laboratorio de Química General de La Universidad Simón Bolívar. Leonarda Carrillo.Investigación Universitaria Multidisciplinaria100% (1)
- Dilemas MoralesDocument4 pagesDilemas Moralesmichael hidalgoNo ratings yet
- 08-2-El Rol Del Auditor Frente A La Ley 8204Document56 pages08-2-El Rol Del Auditor Frente A La Ley 8204Alonso BermúdezNo ratings yet
- 1 Planif y Des Urbano Marco ConceptualDocument47 pages1 Planif y Des Urbano Marco ConceptualmusucanchaNo ratings yet
- Mapa Conceptual El Texto y Su EstructuraDocument1 pageMapa Conceptual El Texto y Su EstructuraMitsuri Diaz Herazo75% (8)
- BannerDocument3 pagesBannerJessica Paola León OrtegaNo ratings yet
- EdafologiaDocument11 pagesEdafologiaMilagros Malpartida PeraunaNo ratings yet
- Tarea Las Partes de La Planta V02Document2 pagesTarea Las Partes de La Planta V02Carlos E SalinasNo ratings yet
- Tarea Academica 4 - Gestión GeneralDocument10 pagesTarea Academica 4 - Gestión GeneralLeonela Villarreal CadilloNo ratings yet
- Medición de Hidrocarburos en El PerúDocument74 pagesMedición de Hidrocarburos en El Perúaltamirano21100% (1)
- Mesa de GuanipaDocument8 pagesMesa de GuanipagianfrancoNo ratings yet
- 3.4 Distribuciones Derivadas Del MuestreoDocument18 pages3.4 Distribuciones Derivadas Del MuestreoJuan DavidNo ratings yet
- 2011 - Clase NI IMAQDocument12 pages2011 - Clase NI IMAQjirlfeNo ratings yet
- La Clínica de Las DiscapacidadesDocument56 pagesLa Clínica de Las DiscapacidadesMartínez AndreaNo ratings yet
- El Arte de La Buena VidaDocument11 pagesEl Arte de La Buena VidaTeólogo Laico33% (3)
- Diseño Factorial Completo Taller 2X2Document15 pagesDiseño Factorial Completo Taller 2X2Rosario CervantesNo ratings yet
- ProblemáticaDocument4 pagesProblemáticaSUSANA CRISTINA CRESPO ARGUDONo ratings yet
- Historia Partida DobleDocument2 pagesHistoria Partida DobleKaren Portal PretelNo ratings yet
- Manual 2 Nivel Reiki Usui-TibetanoDocument7 pagesManual 2 Nivel Reiki Usui-TibetanoMarcela Martha IbañezNo ratings yet
- Variaciones Lingüísticas ListoDocument10 pagesVariaciones Lingüísticas ListoMaría PazNo ratings yet
- Perfil Del Acosador y de La VíctimaDocument5 pagesPerfil Del Acosador y de La VíctimaParalapapiricoipiNo ratings yet
- C# para Programadores de VB6Document90 pagesC# para Programadores de VB6Luis Enrique Stobschinski100% (1)